Voorspelling van transcriptie activator sequenties in het maïsgenoom
22 augustus 2017
UvA-onderzoekers Rurika Oka, Blaise Weber, Maike Stam en Huub Hoefsloot van het Swammerdam Institute for Life Sciences en Franziska Turck en Johan Zicola van het Max Planck Institute for Plant Breeding Research in Keulen, Duitsland publiceerden de resultaten van hun onderzoek werden op in een speciale editie van Genome Biology over genoom-brede planten epigenetica.
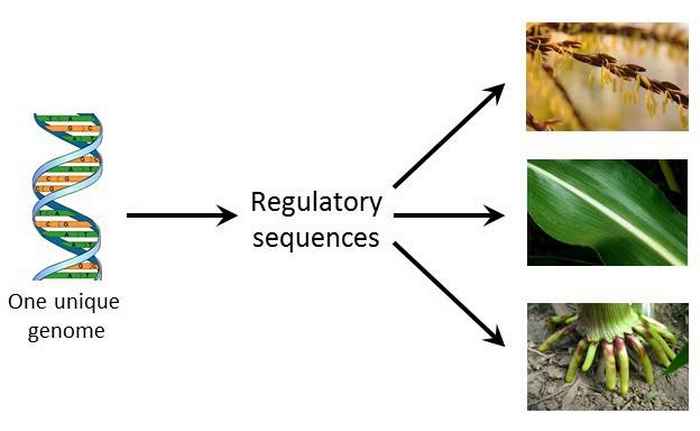
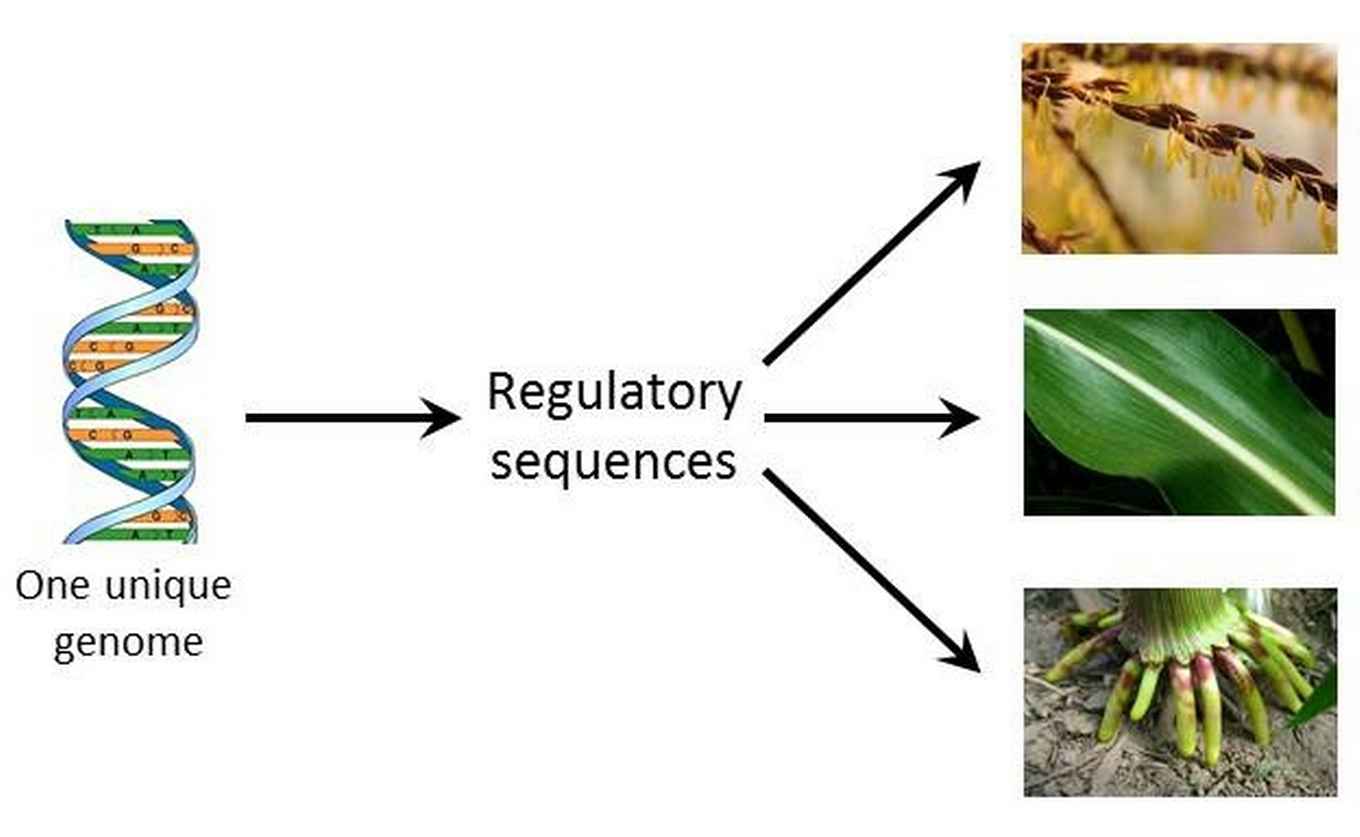
De differentiatie van cellen en hun respons op externe signalen zoals licht, temperatuur en pathogenen wordt grotendeels bepaald door het aan- en uitstaan van regulerende DNA sequenties. Positieve transcriptie regulatoren (activatoren) zijn niet-coderende DNA-sequenties die de transcriptie van genen activeren. Deze regulatoren kunnen zich vlakbij, maar ook ver van de genen die ze reguleren bevinden.
Onontbeerlijk
Kennis over de transcriptie regulerende code is onontbeerlijk voor de functionele annotatie van het genoom en de selectie van gewenste eigenschappen in de plantenveredeling. De meest genoom-brede studies naar activator sequenties worden uitgevoerd op dieren, waardoor hier al duizenden activator sequenties zijn geïdentificeerd.
De regulatie code van planten, met name die van gewasplanten, is echter nog nauwelijks bekend. Een aantal plantenactivator sequenties waren al ontdekt en één voor één experimenteel gekarakteriseerd, maar tot nu toe is genoom-wijde identificatie uitgebleven. Met deze studie wordt een belangrijke stap genomen bij het ontrafelen van de regulatie code van het belangrijke voedingsgewas maïs.
Genoom-brede voorspelling van transcriptie regulatoren in maïs
Uit genoom-brede studies in dieren is ontdekt dat specifieke epigenetische markeringen en chromatine structuren correleren met de activatie en repressie van genen en regulerende DNA sequenties. Bijvoorbeeld, om activatoren bij dieren te identificeren wordt gezocht naar acetylering van het aminozuur lysine op positie 27 van histoneiwit 3 (H3K27ac), in aanwezigheid van lage DNA-methylering en open chromatinegebieden. Uit onderzoek op planten bleek dat voor activator sequenties in planten waarschijnlijk soortgelijke epigenetische kenmerken gelden. Om positieve transcriptie regulatoren in het maïs gewas te voorspellen, hebben de onderzoekers beschikbare genoom-brede DNA-methyleringsgegevens geïntegreerd met nieuw gegenereerde kaarten van de chromatine toegankelijkheid en histoneiwit 3-aminozuur-lysine-9 acetylatie (H3K9ac, ook een activeringskenmerk) in twee verschillende plantenweefsels. Ongeveer 1500 regio's werden geclassificeerd als regulator kandidaten, waaronder drie reeds voorspelde regio’s. Ongeveer de helft van de kandidaten waren meer dan 10.000 baseparen verwijderd van hun vermeende doelgenen. Opvallend was dat de chromatine profielen van deze regulatoren overeenkomsten, maar ook verschillen vertonen met dierlijke regulatoren. Het huidige onderzoek is gericht op de verificatie en verdere karakterisering van de geïdentificeerde kandidaten.Het biedt niet alleen een beter begrip van de genetische regelgeving in planten, maar verruimt ook de gereedschapskist voor de functionele annotatie van complexe plantgenomen, en biedt nieuwe perspectieven voor de plantenveredeling.